Biocódices, junto a otros laboratorios e institutos científicos nacionales, participó durante 2020 del Proyecto Argentino Interinstitucional de genómica de SARS-CoV-2 (PAIS).
El objetivo general del proyecto fue analizar el genoma completo de las cepas del SARS-CoV-2 que circulan en Argentina para estudiar su origen y dispersión en el país, en el contexto de las cepas circulantes en Latinoamérica y el mundo, como así también analizar las mutaciones que pudieran afectar el diagnóstico, la transmisión y la virulencia del virus.
Para ello se realizó un muestreo inicial en todo el país de aproximadamente 1000 genomas de SARS-CoV-2. Las muestras obtenidas fueron secuenciadas por NGS y analizadas desde varios enfoques.
Biocódices fue uno de los nodos de secuenciación y bioinformática. A continuación nos cuentan su experiencia y los objetivos logrados.
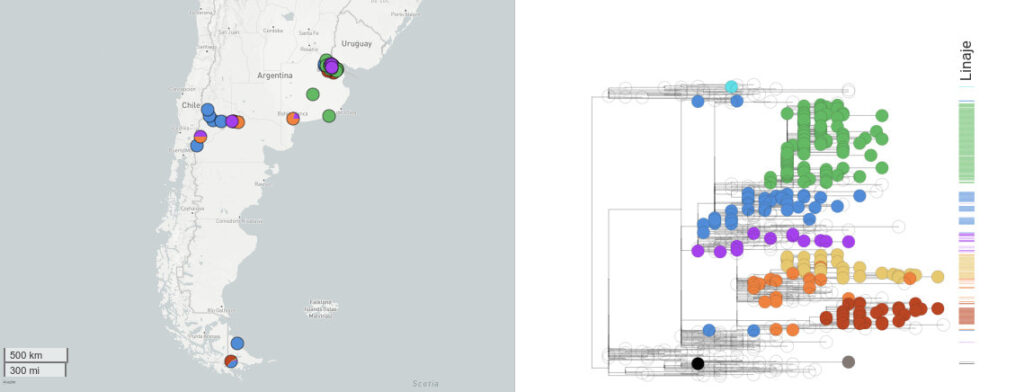
¨Lo que hicimos fue analizar secuencias genómicas de aproximadamente 100 muestras de SARS-COV-2 provenientes de diferentes centros de salud de nuestro país¨ explica Hernán Dopazo director científico. La descripción detallada de las mutaciones en el genoma del virus permitió caracterizar qué linajes del virus se encontraban en el país en ese momento.
«En la secuenciación, obtuvimos el genoma completo del virus mediante el procesamiento de las muestras de ARN colectadas de pacientes infectados¨ comentó Jeremías Zubrzycki director del laboratorio.
¨En bioinformática, colaboramos con la ejecución de un pipeline de análisis y visualización de datos que permitieron entender en detalle el rendimiento de diversos métodos de secuenciación, tipos de librerías genómicas y la distribución de mutaciones de los diferentes linajes de SARS-COV 2 ¨ explicó Juan Manuel Berros, bioinformático y becario de CONICET en empresa que colaboró con este proyecto.

